pour la recherche uniquement
P22077 DUB inhibiteur
Réf. CatalogueS7133
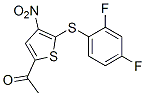
Structure chimique
Poids moléculaire: 315.32
Contrôle Qualité
| Cibles apparentées | Proteasome E1 Activating E3 Ligase p97 SUMO E2 conjugating |
|---|---|
| Autre DUB Inhibiteurs | PR-619 P5091 IU1 b-AP15 ML323 LDN-57444 VLX1570 TCID EOAI3402143 ML364 |
Informations chimiques, stockage et stabilité
| Poids moléculaire | 315.32 | Formule | C12H7F2NO3S2 |
Stockage (À compter de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 1247819-59-5 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | N/A | Smiles | CC(=O)C1=CC(=C(S1)SC2=C(C=C(C=C2)F)F)[N+](=O)[O-] | ||
Solubilité
|
In vitro |
DMSO
: 63 mg/mL
(199.79 mM)
Ethanol : 1 mg/mL Water : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Saisir les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Saisir la formulation in vivo (Ceci est seulement le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouterμL PEG300, mélanger et clarifier, puis ajouterμL Tween 80, mélanger et clarifier, puis ajouter μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouter μL Huile de maïs, mélanger et clarifier.
Note : 1. Veuillez vous assurer que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue, lors de lajout précédent, est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie chaud peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
USP7
(Cell-free assay) 8.6 μM
USP47
8.74 μM(EC50)
|
|---|---|
| In vitro |
P22077 est un analogue de l'inhibiteur de l'USP7 récemment découvert P5091. Ce composé a une activité négligeable contre DEN1 et SENP2core sur la plage de concentrations testée, mais inhibe l'USP7 avec une IC50 de 8 μM. Les activités inhibitrices de cette substance chimique in vitro contre un panel de DUBs, de cystéine protéases et d'autres familles d'enzymes protéolytiques démontrent qu'elle inhibe l'USP7 et la DUB USP47 étroitement apparentée. Elle inhibe un sous-ensemble plus petit de DUBs à une concentration de 15 à 45 mM dans les lysats cellulaires. Ce composé induit la mort cellulaire (tumorale) avec des valeurs d'EC50 dans la plage des faibles micromolaires. Son inhibition a montré des changements dans les niveaux de protéines ubiquitinées qui sont distincts de l'inhibiteur à large spécificité. De plus, la MS quantitative suggère que les composants de l'E3 Ubiquitin ligase RBX1, DCAF7, DCAF11, et la protéine de liaison aux dommages de l'ADN 1 (DDB1) sont réduits après traitement cellulaire avec cette substance chimique. |
| Essai kinase |
Essais UbL-EKL
|
|
Sauf indication contraire, l'isopeptidase recombinante est mélangée avec UbL-EKL et EKLsubstrate I à des concentrations finales de 20, 50 et 20 nM, respectivement, dans un volume total de 100 μL dans un puits d'une plaque à 96 puits à parois noires. Toutes les dilutions sont effectuées dans un tampon d'essai d'isopeptidase (20 mM Tris-HCl, pH 8,0, 2 mM CaCl2 et 2 mM β-mercaptoéthanol). L'augmentation de l'intensité de fluorescence au fil du temps est déterminée sur un lecteur de plaques de fluorescence Perkin Elmer Envision avec des filtres d'excitation et d'émission correspondant au peptide de transfert d'énergie par résonance de fluorescence utilisé. Sauf indication contraire, les unités de fluorescence relatives nettes (RFU) sont déterminées en soustrayant la valeur de RFU du blanc (20 nM EKL substrate I ou 100 nM EKL substrate II dans un tampon d'essai d'isopeptidase) de chaque point de données. Les expériences de sensibilité SENP2core, SUMO3-EKL sont réalisées en mélangeant 0–100 fM de SENP2core avec 50 nM de SUMO3-EKL et 100 nM de EKL substrate I dans un volume total de 100 μL comme ci-dessus.
|
|
| In vivo |
L'inhibition de l'USP7 par P22077 inhibe également la croissance des tumeurs mélanomateuses in vivo. |
Références |
|
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
