pour la recherche uniquement
SRT1720 HCl Sirtuin activateur
Réf. CatalogueS1129
Structure chimique
Poids moléculaire: 506.02
Contrôle Qualité
Produits souvent utilisés avec SRT1720 HCl
| Cibles apparentées | HDAC JAK BET Histone Methyltransferase PKC PARP HIF PRMT EZH2 AMPK |
|---|---|
| Autre Sirtuin Inhibiteurs | Sirtinol Fisetin 3-TYP AGK2 SRT2104 (GSK2245840) OSS_128167 SirReal2 Thiomyristoyl NRD167 SRT2183 |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| CACs | Function Assay | 4 μM | 30 min | DMSO | induces acute SIRT1 activation | 26254104 |
| MC3T3-E1 | Function Assay | 10 µM | 1 h | reduces the TGF-β-stimulated VEGF release in dose- and time-dependent manner | 26136978 | |
| MC3T3-E1 | Function Assay | 10 µM | 12 h | reduces the VEGF mRNA expression levels stimulated by TGF-β | 26136978 | |
| MC3T3-E1 | Function Assay | 20 μM | 1 h | suppresses the TGF-β-induced phosphorylation of p44/p42 MAP kinase or SAPK/JNK | 26136978 | |
| WE-68 | Apoptosis Assay | 0-24 μM | 24 h | induces cell death in dose dependently | 26055805 | |
| SK-ES-1 | Apoptosis Assay | 0-10 μM | 24 h | induces cell death in dose dependently | 26055805 | |
| SK-N-MC | Apoptosis Assay | 0-2.5 μM | 24 h | induces cell death in dose dependently | 26055805 | |
| WE-68 | Function Assay | 20 μM | 0-24 h | activates caspase 3/7 | 26055805 | |
| SK-ES-1 | Function Assay | 10 μM | 0-24 h | activates caspase 3/7 | 26055805 | |
| SK-N-MC | Function Assay | 3 μM | 0-24 h | activates caspase 3/7 | 26055805 | |
| NRK-49F | Function Assay | 0–2 μM | 36 h | increases expression of α-SMA and fibronectin dose dependently | 26022003 | |
| NRK-49F | Function Assay | 0–2 μM | 36 h | enhances phosphorylation of EGFR and PDGFRβ | 26022003 | |
| NRK-49F | Function Assay | 0–2 μM | 36 h | enhances STAT3 phosphorylation | 26022003 | |
| RAW264.7 | Function Assay | 1 μM | 6 h | upregulates the reduced SIRT1 protein or mRNA levels by high glucose | 25793995 | |
| MCF10A | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| MCF-7 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| T47D | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| SKBR3 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| MDA-MB-231 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| SUM149 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| HS578T | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| BT20 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| A459 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| HCT116 | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| Neu | Growth Inhibition Assay | 0-20 μM | 24 h | reduces cell viability dose dependently | 25411356 | |
| MDA-MB-231 | Function Assay | 5 μM | 8 h | increases the number of acidic vesicular organelles | 25411356 | |
| MDA-MB-231 | Function Assay | 5 μM | 16 h | induces lysosomal membrane permeabilization | 25411356 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | suppresses the FGF-2-stimulated osteoprotegerin release | 25290095 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | attenuates the FGF-2-induced osteoprotegerin mRNA expression | 25290095 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | attenuates the FGF-2-induced osteoprotegerin mRNA expression | 25290095 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | suppresses the BMP-4-stimulated VEGF release | 24435444 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | suppresses the PGF2α-stimulated OPG release | 24333336 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | reduces the PGF2α-stimulated phosphorylation of p44/p42 MAP kinase | 24333336 | |
| MC3T3-E1 | Function Assay | 10 μM | 60 min | attenuates the PGF2α-induced phosphorylation of both MEK1/2 and Raf-1 | 24333336 | |
| RPE | Cell Viability Assay | 5 µM | 1 h | attenuates OAβ-induced decrease of cell viability | 24036938 | |
| 9607 | Cell Viability Assay | 1 μM | 36 h | increases the cell viability compared with melatonin alone | 23726949 | |
| 9607 | Function Assay | 1 μM | 36 h | increases SIRT1 and decreased acetylated-p53 expression | 23726949 | |
| RPMI.8226 | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| U266 | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| MM.1S | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| KMS12 | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| LR5 | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| MM.1R | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| Ina6 | Cell Viability Assay | 7/10 μM | 24 h | decreases viability concentration dependently | 21950728 | |
| RPMI-8226 | Apoptosis Assay | 7/10 μM | 24 h | induces a significant increase in the Annexin V+/PI− apoptosis | 21950728 | |
| MM.1R | Apoptosis Assay | 7/10 μM | 24 h | induces a significant increase in the Annexin V+/PI− apoptosis | 21950728 | |
| H411EC3 | Function Assay | 50/100 nM | 6 h | increases SIRT1 activity in the presence of TSA, PEPCK activity, mRNA levels of Pck1 and Pgc1α, and elevating glucose production | 21212096 | |
| hepatocytes | Function Assay | 10 nM | 6 h | increases SIRT1 activity in the presence of TSA, PEPCK activity, mRNA levels of Pck1 and Pgc1α, and elevating glucose production | 21212096 | |
| hepatocytes | Function Assay | 10 nM | 6 h | increases Hmgcr and Acc gene expression | 21212096 | |
| U2OS | Function assay | 0.10 uM | Activation of SIRT1 in human U2OS cells assessed as decrease in p53 deacetylation level at 0.10 uM | 18046409 | ||
| A673 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for A673 cells | 29435139 | |||
| DAOY | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for DAOY cells | 29435139 | |||
| BT-37 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-37 cells | 29435139 | |||
| RD | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for RD cells | 29435139 | |||
| MG 63 (6-TG R) | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for MG 63 (6-TG R) cells | 29435139 | |||
| NB1643 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB1643 cells | 29435139 | |||
| OHS-50 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for OHS-50 cells | 29435139 | |||
| Rh41 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh41 cells | 29435139 | |||
| Rh30 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh30 cells | 29435139 | |||
| LAN-5 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for LAN-5 cells | 29435139 | |||
| Rh18 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh18 cells | 29435139 | |||
| Cliquez pour voir plus de données expérimentales sur la lignée cellulaire | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 506.02 | Formule | C25H23N7OS.HCl |
Stockage (À compter de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 1001645-58-4 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | N/A | Smiles | C1CN(CCN1)CC2=CSC3=NC(=CN23)C4=CC=CC=C4NC(=O)C5=NC6=CC=CC=C6N=C5.Cl | ||
Solubilité
|
In vitro |
DMSO
: 100 mg/mL
(197.62 mM)
Water : Insoluble Ethanol : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Saisir les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Saisir la formulation in vivo (Ceci est seulement le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouterμL PEG300, mélanger et clarifier, puis ajouterμL Tween 80, mélanger et clarifier, puis ajouter μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouter μL Huile de maïs, mélanger et clarifier.
Note : 1. Veuillez vous assurer que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue, lors de lajout précédent, est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie chaud peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
SIRT1
(Cell-free assay) 0.16 μM(EC50)
|
|---|---|
| In vitro |
Le ratio d'activation maximal du SRT1720 par rapport aux homologues sirtuines les plus proches, SIRT2 (EC1,5 = 37 μM) et SIRT3 (EC1,5 > 300 μM), est jusqu'à 781 %. Le SRT1720 se lie au complexe enzyme-peptide substrat SIRT1 à un site allostérique amino-terminal au domaine catalytique et abaisse la constante de Michaelis pour les substrats acétylés. Le SRT1720 pourrait réduire les niveaux de glucose postprandial. Le SRT1720 n'a pas d'effet sur la glycémie à jeun chez les souris nourries avec un régime standard, révélant que l'activation pharmacologique de SIRT1 est peu susceptible d'induire une hypoglycémie. Le SRT1720 réduit significativement l'hyperinsulinémie après 4 semaines, normalisant partiellement les niveaux d'insuline accrus. Le traitement au SRT1720 augmente la capacité mitochondriale de 15 % dans le muscle gastrocnémien, mesurée par l'activité de la citrate synthase. Des concentrations plus élevées de SRT1720 (15 μM) induisent une diminution modeste (10-20 %) de la viabilité des cellules normales. Le SRT1720 inhibe également significativement la migration des cellules de myélome multiple dépendantes du VEGF. |
| Essai kinase |
Essai de polarisation de fluorescence SIRT1
|
|
Dans l'essai FP de SIRT1, l'activité de SIRT1 est surveillée à l'aide d'un peptide de 20 acides aminés (Ac-Glu-Glu-Lys(biotin)-Gly-Gln-Ser-Thr-Ser-Ser-His-Ser-Lys(Ac)-Nle-Ser-Thr-Glu-Gly–Lys(MR121 ou Tamra)-Glu-Glu-NH2) dérivé de la séquence de p53. Le peptide est lié N-terminalement à la biotine et modifié C-terminalement avec un marqueur fluorescent. La réaction pour surveiller l'activité enzymatique est un essai enzymatique couplé où la première réaction est la réaction de désacétylation catalysée par SIRT1 et la seconde réaction est le clivage par la trypsine au niveau du résidu lysine nouvellement exposé. La réaction est arrêtée et la streptavidine est ajoutée afin d'accentuer les différences de masse entre le substrat et le produit. La sensibilité de l'essai FP permet l'identification du SRT1720. Les conditions de réaction de polarisation de fluorescence sont les suivantes : 0,5 μM de substrat peptidique, 150 μM de βNAD+, 0-10 nM de SIRT1, 25 mM de Tris-acétate pH 8, 137 mM de Na-Ac, 2,7 mM de K-Ac, 1 mM de Mg-Ac, 0,05 % de Tween-20, 0,1 % de Pluronic F127, 10 mM de CaCl 2, 5 mM de DTT, 0,025 % de BSA et 0,15 mM de nicotinamide. La réaction est incubée à 37 °C et arrêtée par l'addition de nicotinamide, et la trypsine est ajoutée pour cliver le substrat désacétylé. Cette réaction est incubée à 37 °C en présence de 1 μM de streptavidine. La polarisation fluorescente est déterminée aux longueurs d'onde d'excitation (650 nm) et d'émission (680 nm).
|
|
| In vivo |
Chez les souris DIO, le SRT1720 mime plusieurs des effets observés après une restriction calorique, notamment une amélioration de la sensibilité à l'insuline, la normalisation des taux de glucose et d'insuline, et une augmentation de la capacité mitochondriale. De plus, chez les souris obèses induites par l'alimentation et génétiquement obèses, le SRT1720 améliore la sensibilité à l'insuline, abaisse la glycémie plasmatique et augmente la capacité mitochondriale. Ainsi, le SRT1720 est un nouvel agent thérapeutique prometteur pour le traitement des maladies liées au vieillissement telles que le diabète de type 2. Conformément à une tolérance au glucose améliorée, le débit de perfusion de glucose nécessaire pour maintenir l'euglycémie est environ 35 % plus élevé chez les rats fa/fa traités au SRT1720, et le taux total d'élimination du glucose est augmenté d'environ 20 %. Le SRT1720 prévient également la croissance tumorale du myélome multiple. |
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
| Western blot | Cleaved-PARP-1 / Cleaved-caspase-3 / LC3-II / p62 / SIRT1 |
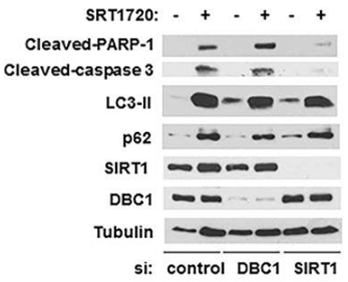
|
26655844 |
| Immunofluorescence | Cathepsin B |
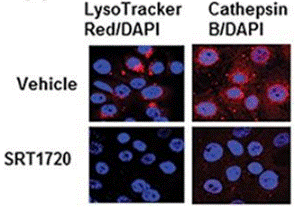
|
26655844 |
| Growth inhibition assay | Cell viability |

|
25411356 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
Foire aux questions
Question 1:
How can we prepare it for in vivo mouse studies?
Réponse :
It can be dissolved in 30% PEG 400+0.5% Tween 80+5% Propylene glycol at 30mg/ml as a suspension, which is fine for oral gavage. We’ve also found that it can be dissolved in 2% DMSO+30% PEG 300+1% Tween 80+ddH2O at 3mg/ml clearly, making it suitable for injection. When preparing the solution, please dissolve the compound in DMSO clearly first, then add PEG and Tween. After they are mixed well, dilute with water.
