pour la recherche uniquement
GSK126 inhibiteur de l'EZH2
Réf. CatalogueS7061
Structure chimique
Poids moléculaire: 526.67
Contrôle Qualité
| Cibles apparentées | HDAC JAK BET Histone Methyltransferase PKC PARP HIF PRMT AMPK Histone Acetyltransferase |
|---|---|
| Autre EZH2 Inhibiteurs | PF-06821497 Tulmimetostat (CPI-0209) Valemetostat (DS-3201) GSK343 Lirametostat (CPI-1205) EBI-2511 EZH2/HSP90-IN-29 SHR2554 |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| human U87MG cells | Cytotoxic assay | 72 h | Cytotoxicity against human U87MG cells assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=28.5 μM. | 24767850 | ||
| human A549 cells | Cytotoxic assay | 72 h | Cytotoxicity against human A549 cells assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=18.7 μM. | 24767850 | ||
| human T98G cells | Cytotoxic assay | 72 h | Cytotoxicity against human T98G cells assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=12.6 μM. | 24767850 | ||
| human Daudi cells | Cytotoxic assay | 72 h | Cytotoxicity against human Daudi cells assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=11.2 μM. | 24767850 | ||
| human PC3 cells | Cytotoxic assay | 72 h | Cytotoxicity against human PC3 cells assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=9.4 μM. | 24767850 | ||
| human U2932 cells | Cytotoxic assay | 72 h | Cytotoxicity against human U2932 cells assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=6.7 μM. | 24767850 | ||
| human HeLa cells | Function assay | 72 h | Inhibition of EZH2 in human HeLa cells assessed as reduction in H3K27me3 levels incubated for 72 hrs by ELISA method, IC50=0.28 μM. | 26189078 | ||
| human Pfeiffer cells | Cytotoxic assay | 72 h | Cytotoxicity against human Pfeiffer cells expressing EZH2 A667G mutant assessed as growth inhibition after 72 hrs by WST-1 assay, GI50=0.18 μM. | 24767850 | ||
| infected SF9 cells | Binding affinity to EZH2 (unknown origin) expressed in baculovirus infected SF9 cells co-expressing SUZ12/EED/RbAp48 complex assessed as binding off-rate at 0.4 uM incubated for 20 mins by Q-TOF mass spectrometry | 27512831 | ||||
| A673 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for A673 cells | 29435139 | ||||
| DAOY cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for DAOY cells | 29435139 | ||||
| Saos-2 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Saos-2 cells | 29435139 | ||||
| BT-37 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-37 cells | 29435139 | ||||
| RD cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for RD cells | 29435139 | ||||
| SK-N-SH cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-SH cells | 29435139 | ||||
| BT-12 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-12 cells | 29435139 | ||||
| MG 63 (6-TG R) cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for MG 63 (6-TG R) cells | 29435139 | ||||
| NB1643 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB1643 cells | 29435139 | ||||
| OHS-50 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for OHS-50 cells | 29435139 | ||||
| Rh41 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh41 cells | 29435139 | ||||
| SK-N-MC cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-MC cells | 29435139 | ||||
| LAN-5 cells | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for LAN-5 cells | 29435139 | ||||
| Cliquez pour voir plus de données expérimentales sur la lignée cellulaire | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 526.67 | Formule | C31H38N6O2 |
Stockage (À compter de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 1346574-57-9 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | GSK2816126A, GSK2816126 | Smiles | CCC(C)N1C=C(C2=C(C=C(C=C21)C3=CN=C(C=C3)N4CCNCC4)C(=O)NCC5=C(C=C(NC5=O)C)C)C | ||
Solubilité
|
In vitro |
DMSO
: 5 mg/mL
(9.49 mM)
Ethanol : 4 mg/mL Water : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Saisir les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Saisir la formulation in vivo (Ceci est seulement le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouterμL PEG300, mélanger et clarifier, puis ajouterμL Tween 80, mélanger et clarifier, puis ajouter μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouter μL Huile de maïs, mélanger et clarifier.
Note : 1. Veuillez vous assurer que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue, lors de lajout précédent, est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie chaud peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
EZH2
(Cell-free assay) 9.9 nM
|
|---|---|
| In vitro |
In vitro, GSK126 inhibe le plus puissamment H3K27me3, suivi de H3K27me2 dans les lignées cellulaires DLBCL de type sauvage et mutantes d'EZH2. Ce composé inhibe également efficacement la prolifération des lignées cellulaires DLBCL mutantes d'EZH2 et induit l'activation transcriptionnelle des gènes cibles d'EZH2 dans les lignées cellulaires sensibles. Dans les cellules mutantes A687V EZH2, ce traitement entraîne une diminution globale de H3K27me3, une activation génique robuste, une activation de la caspase et une diminution de la prolifération. Dans les cellules H2087 parentales, il inhibe l'expression de VEGF-A et de Ser(473)-AKT phosphorylée, et provoque ainsi l'inhibition de la prolifération, de la migration et de la métastase des cellules.
|
| Essai kinase |
Test EZH2
|
|
Le complexe PRC2 à cinq membres (Flag–EZH2, EED, SUZ12, AEBP2, RbAp48) contenant soit l'EZH2 de type sauvage, soit l'EZH2 mutante est préparé. GSK126 est dissous dans le DMSO et testé à des concentrations de 0,6 nM à 300 nM avec une concentration finale de DMSO de 2,5 %. Contrairement à l'EZH2 de type sauvage qui préfère H3K27me0 comme substrat in vitro, les mutants Y641 d'EZH2 préfèrent H3K27me2 et ont peu d'activité avec H3K27me0 ou H3K27me1. Le mutant A677G est distinct des formes sauvage et mutantes Y641 d'EZH2 en ce qu'il méthyle efficacement H3K27me0, H3K27me1 et H3K27me2 ; par conséquent, des peptides d'histone H3 (résidus 21–44 ; 10 μM final) avec K27me0 (type sauvage, A677G EZH2), K27me1 (A677G EZH2) ou K27me2 (A677G, Y641N, Y641C, Y641H, Y641S et Y641F EZH2) sont utilisés comme substrats de méthyltransférase. Ce composé est ajouté aux plaques, suivi de l'ajout de 6 nM de complexe EZH2 et de peptide. Comme la puissance de ce produit chimique est proche ou égale à la limite de liaison étroite d'un essai réalisé à [SAM] = Km, les valeurs d'IC50 sont mesurées à une concentration élevée du substrat compétitif SAM par rapport à son Km (7,5 μM SAM, le Km du SAM étant de 0,3 μM). Dans ces conditions, la contribution de la concentration enzymatique devient relativement faible et des estimations précises de Ki peuvent être calculées. Les réactions sont initiées avec [3H]-SAM, incubées pendant 30 min, stoppées par l'ajout d'un excès 500 fois supérieur de SAM non marqué, et le peptide produit méthylé est capturé sur des filtres de phosphocellulose selon le protocole fourni par le fournisseur pour les plaques MSPH Multiscreen. Les plaques sont lues sur un TopCount après l'ajout de 20 μL de cocktail Microscint-20. Les valeurs apparentes de Ki sont calculées en utilisant la relation de Cheng–Prusoff pour un inhibiteur compétitif. IC50=Ki (1+[S]/Km)+[E]/2, où E est l'enzyme et S est le substrat.
|
|
| In vivo |
Chez des souris porteuses de xénogreffes KARPAS-422 et Pfeiffer, le GSK126 (150 mg/kg/j, i.p.) diminue le H3K27me3 global, augmente l'expression génique et provoque ainsi une régression tumorale marquée.
|
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
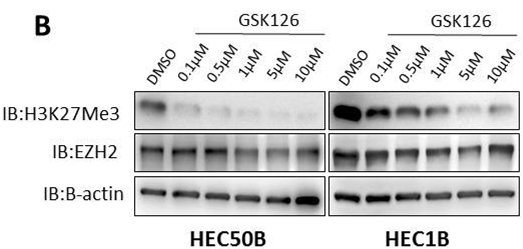
| Western blot | H3K27Me3 / EZH2 XIAP / Survivin / MCL-1 / BID / BIM / BAX / BCL-xl/ Bcl-2 β-catenin / c-Myc / LEF1 / DVL2 / DVL3 / p-GSK3β |
|
28418882 |
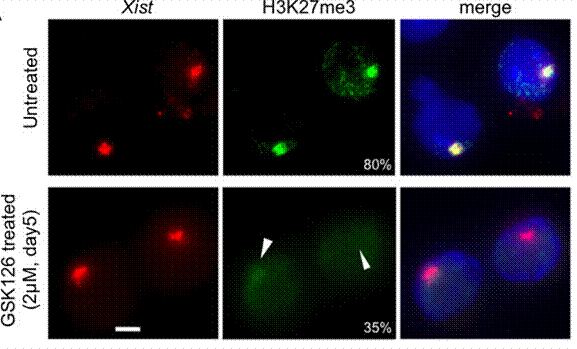
| Immunofluorescence | H3K27me3 |
|
25053977 |
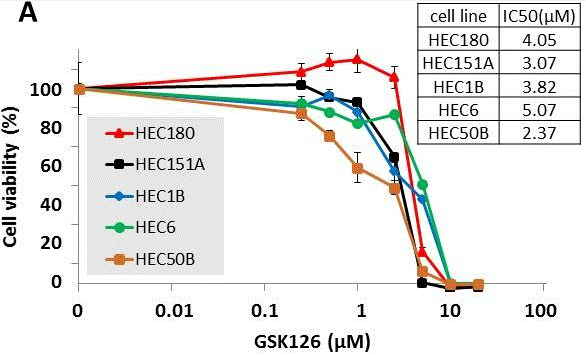
| Growth inhibition assay | Cell viability Cell proliferation |
|
28418882 |
Informations sur lessai clinique
(données du https://clinicaltrials.gov, mis à jour le 2024-05-22)
| Numéro NCT | Recrutement | Conditions | Promoteur/Collaborateurs | Date de début | Phases |
|---|---|---|---|---|---|
| NCT02082977 | Terminated | Cancer|Neoplasms |
GlaxoSmithKline |
April 24 2014 | Phase 1 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
Foire aux questions
Question 1:
Could you please suggest a vehicle for in vivo uses of this compound without oil?
Réponse :
It could be dissolved in 4% DMSO+30% PEG 300+ddH2O (0.5mg/ml).
Question 2:
Does this compound require an activation step to be functional? For example, an acidic or basic environment.
Réponse :
It does not require an activation step to be functional.
