pour la recherche uniquement
Refametinib (RDEA119) MEK inhibiteur
Réf. CatalogueS1089
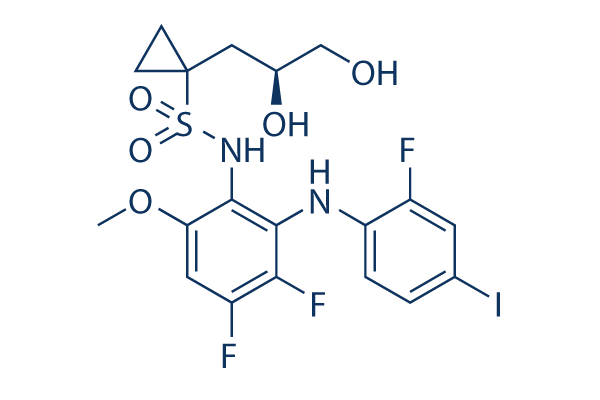
Structure chimique
Poids moléculaire: 572.34
Contrôle Qualité
| Cibles apparentées | ERK p38 MAPK Raf JNK Ras KRas S6 Kinase MAP4K TAK1 Mixed Lineage Kinase |
|---|---|
| Autre MEK Inhibiteurs | PD0325901 (Mirdametinib) U0126-EtOH PD 98059 PD184352 (CI-1040) BIX 02189 Pimasertib (AS-703026) TAK-733 AZD8330 SL-327 BIX 02188 |
Informations chimiques, stockage et stabilité
| Poids moléculaire | 572.34 | Formule | C19H20F3IN2O5S |
Stockage (À compter de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 923032-37-5 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | Bay 86-9766 | Smiles | COC1=CC(=C(C(=C1NS(=O)(=O)C2(CC2)CC(CO)O)NC3=C(C=C(C=C3)I)F)F)F | ||
Solubilité
|
In vitro |
DMSO
: 100 mg/mL
(174.72 mM)
Ethanol : 100 mg/mL Water : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Saisir les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Saisir la formulation in vivo (Ceci est seulement le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouterμL PEG300, mélanger et clarifier, puis ajouterμL Tween 80, mélanger et clarifier, puis ajouter μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, puis ajouter μL Huile de maïs, mélanger et clarifier.
Note : 1. Veuillez vous assurer que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue, lors de lajout précédent, est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie chaud peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
MEK1
(Cell-free assay) 19 nM
MEK2
(Cell-free assay) 47 nM
|
|---|---|
| In vitro |
Refametinib (RDEA119) se lie sélectivement et directement à une poche allostérique des enzymes MEK1/2, et est très efficace pour inhiber la prolifération cellulaire dans plusieurs lignées de cellules tumorales, y compris A375, SK-MEI-28, Colo205, HT-29 et BxPC3. Il inhibe la croissance dépendante de l'ancrage de lignées de cellules cancéreuses humaines abritant la mutation V600E BRAF avec gain de fonction, avec des valeurs de GI50 allant de 67 à 89 nM. Dans des conditions indépendantes de l'ancrage, les valeurs de GI50 pour toutes les lignées cellulaires testées sont similaires (40-84 nM). Ce composé montre une sélectivité tissulaire qui réduit son potentiel d'effets secondaires liés au système nerveux central. Il inhibe puissamment la prolifération des 4 lignées cellulaires qui abritaient la mutation BRAF, mais a des effets nuls ou modestes sur les 4 autres cellules qui abritaient le BRAF de type sauvage (IC50 de 0,034-0,217 μM contre 1,413-34,120 μM). L'effet inhibiteur de Refametinib dans les lignées cellulaires sélectionnées OCUT1 (BRAF V600E(+), PIK3CA H1047R(+)) et SW1376 (BRAF V600E(+)) est amélioré par la combinaison avec l'inhibiteur de mTOR, le temsirolimus. Le Refametinib et le temsirolimus montrent également des effets synergiques sur la mort autophagique des cellules OCUT1 et KAT18 testées sélectivement.
|
| Essai kinase |
Essai kinase MEK
|
|
La kinase ERK2 murine (mERK2) K52A/T183A inactive est purifiée par affinité à partir d'Escherichia coli exprimée à l'aide du vecteur pET21a. L'activité de la MEK1 kinase est déterminée en utilisant mERK2 K52A T183A comme substrat. L'enzyme MEK1 recombinante (5 nM) est d'abord activée par 0,02 unité ou 1,5 nM de RAF1 en présence de 25 mM HEPES (pH 7,8), 1 mM MgCl2, 50 mM NaCl, 0,2 mM EDTA et 50 μM ATP pendant 30 minutes à 25 °C. Les réactions sont initiées par l'ajout de 2 μM de mERK2K52A T183A et 2,5 μCi [γ-33P] ATP dans un volume total de 20 μL. L'activité de la MEK2 kinase est déterminée de manière similaire, sauf que l'activation par RAF1 n'est pas nécessaire et que 11 nM d'enzyme MEK2 (active) sont utilisés dans les essais. Le profilage des kinases pour Refametinib (RDEA119) est réalisé par Invitrogen en utilisant leur service Select Screen Kinase Profiling. L'essai biochimique Z'-LYTE est utilisé. Ce composé est testé en quadruplicata à 10 μM contre 205 kinases.
|
|
| In vivo |
L'administration orale de RDEA119 à 50 mg/kg selon un schéma quotidien unique × 14 conduit à une inhibition de 68 % de la croissance tumorale (TGI) dans le modèle tumoral de mélanome humain A375. Ce composé à 25 mg/kg selon un schéma quotidien unique × 14 entraîne une TGI de 123 % dans le modèle tumoral de carcinome colorectal humain Colo205 (une TGI > 100 % se produit lorsque la tumeur rétrécit en dessous de son volume initial). Une dose de 25 mg/kg une fois par jour × 14 produit une TGI de 56 % et 67 % pour les tumeurs HT-29 et A431, respectivement.
|
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
| Growth inhibition assay | IC50 |
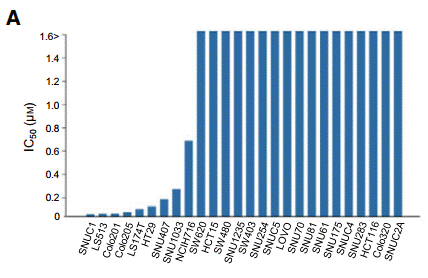
|
29896883 |
| Western blot | p-BRAF / BRAF / p-MEK / MEK / p-ERK / ERK Cyclin D1 / Cyclin E / p27 p-STAT3 / STAT3 |
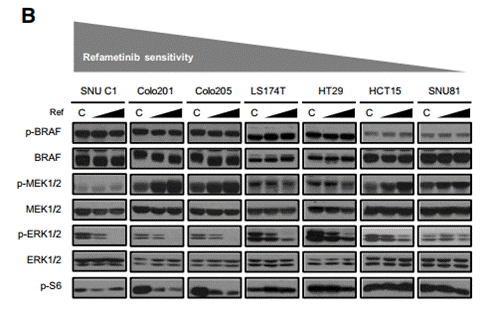
|
29896883 |
Informations sur lessai clinique
(données du https://clinicaltrials.gov, mis à jour le 2024-05-22)
| Numéro NCT | Recrutement | Conditions | Promoteur/Collaborateurs | Date de début | Phases |
|---|---|---|---|---|---|
| NCT01392521 | Completed | Neoplasms |
Bayer |
July 2011 | Phase 1 |
| NCT00785226 | Completed | Advanced Cancer |
Bayer |
November 2008 | Phase 1|Phase 2 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
